基于NGS的微生物检测数据分析方法与技术探究
来源:985论文网 添加时间:2020-01-02 14:42
摘要:高通量测序技术的发展正在逐渐改变诸多生物学领域的研究方法。为应对突发疫情以及新发未知微生物威胁的需求,微生物鉴定技术逐渐从传统的物理化学方法及核酸杂交等分子水平方法进一步走向利用无需培养的测序数据进行快速分析检测。随之而来的是对高通量数据分析在精度及速度的要求。基于高通量测序数据的微生物检测数据分析方法在近些年得到了快速的发展。本文分析了目前基于高通量测序数据的微生物检测数据分析方法,对其数据分析的处理流程和计算方法进行了研究,比较了各个微生物检测数据分析方法的特点及适用场景。最后结合本实验室工作总结微生物检测数据分析方法在实际应用中可能遇到的问题,希望对该应用领域的研究有一定的参考意义。
关键词:高通量测序,微生物检测,数据分析方法,性能评测。
微生物检测旨在通过传统生化、免疫实验方法或者高通量测序方法鉴别宏基因组样品中的微生物种类和定量信息。常用传统微生物检测方法包括涂片镜检法、PCR 扩增法以及基因芯片法等。涂片镜检法通过对样本微生物进行染色,观察大小形态,与图例进行对比从而完成检测工作。其优势为快速、成本低和不需要特殊仪器[1].基因芯片法通过荧光标记探针杂交显示特异微生物的序列信息和位置。其优势为敏感度高、检测快速[2].PCR扩增法利用寡核苷酸引物引导待测基因片段进行扩增,从而能够有效增强检测信号,提高检测效率[3].但是传统微生物检测方法都难以解决未知微生物的检测问题:首先,如果样本中存在未知微生物,则无法通过形态学特征等获取其种属信息,分离培养法和涂片镜检法不再适用;其次,PCR 扩增法和基因芯片技术的探针设计需要对样本的先验知识有所了解,同样也难以鉴别未知微生物[4].
快速准确地检测宏基因组样本中的微生物,确定其种源、毒力等信息是疾病防控和生物安全的关键问题[5].新发未知的微生物由于其突发性、无法获取先验知识等特征,难以通过传统微生物检测方法进行快速、有效的应对。第二代测序(next-generation sequencing,NGS)技术经过 10 年左右的快速发展,功能不断完善,成本逐渐降低,一次运行可以测定千万级别的短序列[6].基于 NGS 的微生物检测的完整流程包括:通过对宏基因组样本进行大规模完全测序,得到宏基因组的核酸序列;再利用生物信息学工具对核酸序列数据进行分析,从而进一步得到微生物基因、耐药性、毒力信息等[7].基于 NGS 的微生物检测技术无需进行需要先验知识的样本形态学特征查找或者探针设计,能够对未知致病微生物进行检测,弥补了传统微生物检测方法的不足,成为预防未知生物威胁的重要手段。
随着测序实验技术的日趋成熟,数据分析方法逐渐成为制约基于 NGS 的微生物检测应用的关键环节。本文对近些年来基于 NGS 的微生物检测数据分析方法的流程设计和关键算法做简要介绍,比较各个数据分析方法的特点及适用情况。最后总结面向应用需求的微生物检测数据分析方法,对相关领域的未来走向提出了预测。希望本文对应用高通量测序技术进行微生物检测的工作人员在数据分析方面提供有价值的参考。
1基于NGS的微生物检测数据分析策略。
根据样本来源、提取方法、建库策略的不同,基于NGS的微生物检测数据分析方法所采取的数据处理策略也略有不同。微生物样本多来自血液、口腔、痰液、病理组织等,在进行DNA或RNA的提取后,利用高通量测序仪建库测序,即获得原始reads数据,存储格式通常为fasta或fastq文件,后者包含碱基质量信息。测序原始数据需要进一步的数据分析以获得其中微生物种群的相关信息[8].
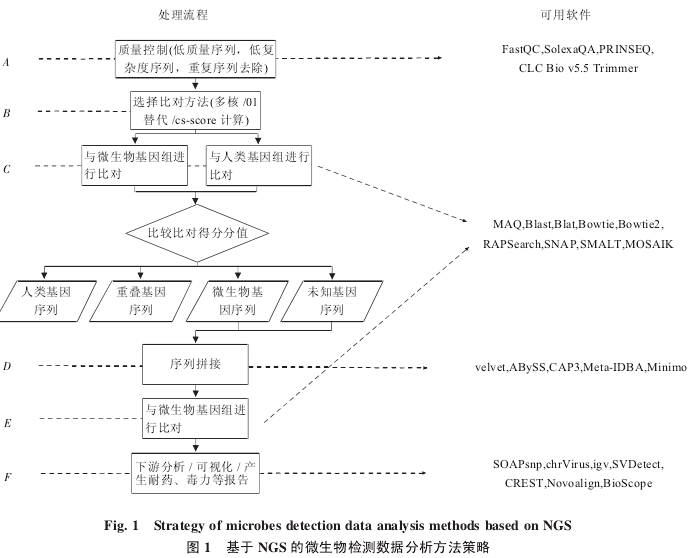
基于NGS的微生物检测数据分析方法面临以下几个关键问题:第一,检测数据分析方法速度要快。由于 NGS 产出数据的通量越来越高,检测数据分析方法的速度需要与之相匹配,才能够达到快速确认,快速应对的目的[9].第二,检测数据分析方法精度要高。面向未知微生物的检测数据分析方法需要尽可能降低检测的假阳性和假阴性比例。如果假阳性过高,无法有效确认疑似微生物,会造成时间和效率的浪费;如果假阴性过高,则无法充分检测出宏基因组样品中的微生物序列,影响后期确认和毒力评估[10].另外,NGS测序仪的测序读长通常为35 ~250 bp (碱基),比第一代测序的读长(650~800 bp)短,因此需要进行de-novo拼接以获得完整微生物序列信息[11].除此之外微生物检测数据分析方法还会遇到计算性能上的问题,需要提高检测方法在不同体系结构上的运行效率,或通过算法优化检测方法所需的计算量等。针对这些问题,目前多数基于NGS的微生物检测方法可以归纳为以下数据分析策略(图 1)。
关键词:高通量测序,微生物检测,数据分析方法,性能评测。
微生物检测旨在通过传统生化、免疫实验方法或者高通量测序方法鉴别宏基因组样品中的微生物种类和定量信息。常用传统微生物检测方法包括涂片镜检法、PCR 扩增法以及基因芯片法等。涂片镜检法通过对样本微生物进行染色,观察大小形态,与图例进行对比从而完成检测工作。其优势为快速、成本低和不需要特殊仪器[1].基因芯片法通过荧光标记探针杂交显示特异微生物的序列信息和位置。其优势为敏感度高、检测快速[2].PCR扩增法利用寡核苷酸引物引导待测基因片段进行扩增,从而能够有效增强检测信号,提高检测效率[3].但是传统微生物检测方法都难以解决未知微生物的检测问题:首先,如果样本中存在未知微生物,则无法通过形态学特征等获取其种属信息,分离培养法和涂片镜检法不再适用;其次,PCR 扩增法和基因芯片技术的探针设计需要对样本的先验知识有所了解,同样也难以鉴别未知微生物[4].
快速准确地检测宏基因组样本中的微生物,确定其种源、毒力等信息是疾病防控和生物安全的关键问题[5].新发未知的微生物由于其突发性、无法获取先验知识等特征,难以通过传统微生物检测方法进行快速、有效的应对。第二代测序(next-generation sequencing,NGS)技术经过 10 年左右的快速发展,功能不断完善,成本逐渐降低,一次运行可以测定千万级别的短序列[6].基于 NGS 的微生物检测的完整流程包括:通过对宏基因组样本进行大规模完全测序,得到宏基因组的核酸序列;再利用生物信息学工具对核酸序列数据进行分析,从而进一步得到微生物基因、耐药性、毒力信息等[7].基于 NGS 的微生物检测技术无需进行需要先验知识的样本形态学特征查找或者探针设计,能够对未知致病微生物进行检测,弥补了传统微生物检测方法的不足,成为预防未知生物威胁的重要手段。
随着测序实验技术的日趋成熟,数据分析方法逐渐成为制约基于 NGS 的微生物检测应用的关键环节。本文对近些年来基于 NGS 的微生物检测数据分析方法的流程设计和关键算法做简要介绍,比较各个数据分析方法的特点及适用情况。最后总结面向应用需求的微生物检测数据分析方法,对相关领域的未来走向提出了预测。希望本文对应用高通量测序技术进行微生物检测的工作人员在数据分析方面提供有价值的参考。
1基于NGS的微生物检测数据分析策略。
根据样本来源、提取方法、建库策略的不同,基于NGS的微生物检测数据分析方法所采取的数据处理策略也略有不同。微生物样本多来自血液、口腔、痰液、病理组织等,在进行DNA或RNA的提取后,利用高通量测序仪建库测序,即获得原始reads数据,存储格式通常为fasta或fastq文件,后者包含碱基质量信息。测序原始数据需要进一步的数据分析以获得其中微生物种群的相关信息[8].
基于NGS的微生物检测数据分析方法面临以下几个关键问题:第一,检测数据分析方法速度要快。由于 NGS 产出数据的通量越来越高,检测数据分析方法的速度需要与之相匹配,才能够达到快速确认,快速应对的目的[9].第二,检测数据分析方法精度要高。面向未知微生物的检测数据分析方法需要尽可能降低检测的假阳性和假阴性比例。如果假阳性过高,无法有效确认疑似微生物,会造成时间和效率的浪费;如果假阴性过高,则无法充分检测出宏基因组样品中的微生物序列,影响后期确认和毒力评估[10].另外,NGS测序仪的测序读长通常为35 ~250 bp (碱基),比第一代测序的读长(650~800 bp)短,因此需要进行de-novo拼接以获得完整微生物序列信息[11].除此之外微生物检测数据分析方法还会遇到计算性能上的问题,需要提高检测方法在不同体系结构上的运行效率,或通过算法优化检测方法所需的计算量等。针对这些问题,目前多数基于NGS的微生物检测方法可以归纳为以下数据分析策略(图 1)。

为了对目前基于 NGS 的微生物检测数据分析方法进行全面评估,我们将分析系统简化为 6 个核心环节,如图 1 所示,分别为:质量控制(A)、比对方法优化(B)、与参考基因组进行比对(C)、序列拼接(D)、与微生物基因组进行比对(E)、下游分析(F)。其中质量控制(环节 A)属于提高微生物检测精度的方法,通常 NGS 产生的测序数据会存在低质量序列、低复杂度序列等,会影响后续分析。因此需要通过质量控制软件对样本测序数据进行处理。比对方法优化(环节 B)属于提高微生物检测速度的方法,由于 NGS 产生上百万片段,数据分析在单个节点上的计算时间可能需要数周,因此需要采用设计算法的查询和匹配效率,以降低数据分析所需的运算量,在数据分析环节上提高速度。序列比对(环节C,E)为微生物检测的核心步骤[12],将样本数据 和 人 类 参 考 基 因 组(human reference geneset,HRG)或 微 生 物 参 考 基 因 组(microbe referencegeneset,MRG)进行比对,清除与HRG比对成功的序列,保留与MRG比对成功的序列,最后获得已知和未知的微生物基因。序列拼接(环节D)将检测出的微生物基因短reads拼接成完整微生物基因序列,从而进行下游分析(环节 F),如可视化[13]、单核苷酸多态性(single nucleotide polymorphisms,SNP)分析[14]等,才能获得基因序列的全部信息,完成微生物检测的整个流程。
2基于NGS的微生物检测数据分析方法。
从2011年开始,基于NGS的微生物检测数据分析方法得到了快速的发展。目前已经发表的数据分析方法包括PathSeq[15]、RINS[16]、CAPSID[17]、VirusSeq[18]、 VirusFinder[19]、 READSCAN[20]、Kraken[21]、SURPI[22]、RIEMS[23]、Pathosphere.org[24]、CS-SCORE[25]、VERSE[26]和VIP[27]等,如表1所示。
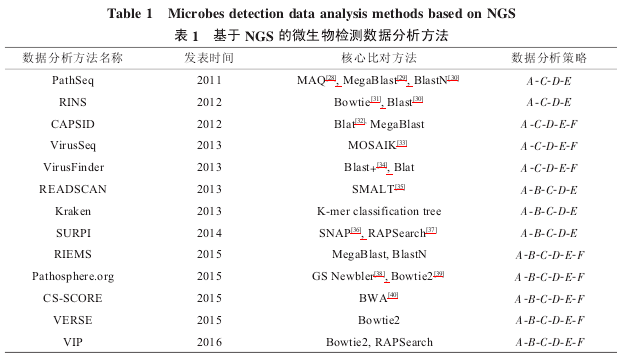 参照前文提到的数据分析策略,可以发现不同的微生物检测计算分析方法主要体现在比对方法优化(环节 B)以及下游分析(环节 F)。早期的数据分析方法如PathSeq、RINS 等,是最初微生物检测数据分析的典型解决方案,并未专门对比对方法进行优化,也缺少下游分析的步骤,属于“基础型”检测方法。而 VirusSeq、VirusFinder、CaPSID 等,在“基础型”的基础上,增加了如病毒结合位点分析等下游分析(环节 F),其检测功能得到了完善,称为“功能型”检测方法。而 READSCAN、Kraken、CS-SCORE在“基础型”的基础上,增加了比对方法优化(环节 B),其处理速度大大增加,属于“速度型”检测方法。近几年出现的 SURPI、RIEMS、Pathosphere.org、VERSE 和 VIP,同时包含比对方法优化和下游分析,能够提供完整的分析流程,属于“完整型”检测方法。
参照前文提到的数据分析策略,可以发现不同的微生物检测计算分析方法主要体现在比对方法优化(环节 B)以及下游分析(环节 F)。早期的数据分析方法如PathSeq、RINS 等,是最初微生物检测数据分析的典型解决方案,并未专门对比对方法进行优化,也缺少下游分析的步骤,属于“基础型”检测方法。而 VirusSeq、VirusFinder、CaPSID 等,在“基础型”的基础上,增加了如病毒结合位点分析等下游分析(环节 F),其检测功能得到了完善,称为“功能型”检测方法。而 READSCAN、Kraken、CS-SCORE在“基础型”的基础上,增加了比对方法优化(环节 B),其处理速度大大增加,属于“速度型”检测方法。近几年出现的 SURPI、RIEMS、Pathosphere.org、VERSE 和 VIP,同时包含比对方法优化和下游分析,能够提供完整的分析流程,属于“完整型”检测方法。
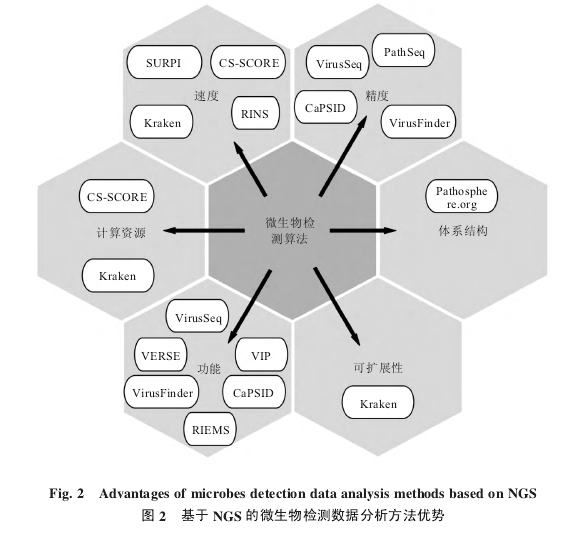
除了数据分析策略带来的基础特点外,由于采用了不同的软件、处理流程等,不同的基于 NGS的微生物检测数据分析方法还有着不同的优势,如图2 所示。
2基于NGS的微生物检测数据分析方法。
从2011年开始,基于NGS的微生物检测数据分析方法得到了快速的发展。目前已经发表的数据分析方法包括PathSeq[15]、RINS[16]、CAPSID[17]、VirusSeq[18]、 VirusFinder[19]、 READSCAN[20]、Kraken[21]、SURPI[22]、RIEMS[23]、Pathosphere.org[24]、CS-SCORE[25]、VERSE[26]和VIP[27]等,如表1所示。

除了数据分析策略带来的基础特点外,由于采用了不同的软件、处理流程等,不同的基于 NGS的微生物检测数据分析方法还有着不同的优势,如图2 所示。

重要提示:转载本站信息须注明来源:985论文网,具体权责及声明请参阅网站声明。
阅读提示:请自行判断信息的真实性及观点的正误,本站概不负责。
论文服务


 专科论文咨询
专科论文咨询
